Processe dados genéticos de microrganismos, obtidos por NGS, e analise-os com um toolkit completo e intuitivo.

- Estamos atuando no mercado desde 2021
- Mais de 4 mil amostras processadas por ano.
- Mais de 7 mil análises realizadas.
- Mais de 17 mil organismos diferentes identificados.
Onde o Varsmetagen atua?
Viroma
Viroma é um tipo de análise de metagenômica realizada em dados de sequenciamento de amostras clínicas ou ambientais, onde o objetivo é a identificação e caracterização dos agentes virais presentes na amostra. Com o Varsmetagen, é possível inspecionar métricas de qualidade, identificar patógenos virais, obter representações gráficas de abundância e diversidade da microbiota (vírus, bactérias e fungos), recuperar genomas virais completos e realizar análises pareadas para laudamento e geração de relatórios.
Microbioma
O pipeline de Microbioma (Metataxonomia ou Metabarcoding) oferece um tipo de análise para Amplicon (16S e ITS) realizada em dados de sequenciamento de amostras clínicas ou ambientais, onde o objetivo é avaliar a diversidade de microrganismos e identificação de disbioses presente na amostra. Com o Varsmetagen, é possível comparar os resultados com o banco de referência do Human Microbiome Project (HMP), além de inspecionar métricas de qualidade, observar disbioses e obter representações gráficas de abundância e diversidade do microbioma (bactérias e fungos).
Metagenômica
O pipeline de bioinformática para metagenomas possibilita a detecção de diversos patógenos (bactérias, vírus, fungos e parasitas eucarióticos) e também de microrganismos simbióticos, por meio de análises taxonômicas de reads e contigs, recuperação de genomas de metagenomas (MAGs) e mapeamento direcionado em genoma de referência. Com o Varsmetagen é possível realizar o diagnóstico de doenças infecciosas, caracterizar o microbioma e micobioma, e emitir laudos e relatórios completos sobre os resultados.
Viroma
Viroma é um tipo de análise de metagenômica realizada em dados de sequenciamento de amostras clínicas ou ambientais, onde o objetivo é a identificação e caracterização dos agentes virais presentes na amostra. Com o Varsmetagen, é possível inspecionar métricas de qualidade, identificar patógenos virais, obter representações gráficas de abundância e diversidade da microbiota (vírus, bactérias e fungos), recuperar genomas virais completos e realizar análises pareadas para laudamento e geração de relatórios.
Microbioma
O pipeline de Microbioma (Metataxonomia ou Metabarcoding) oferece um tipo de análise para Amplicon (16S e ITS) realizada em dados de sequenciamento de amostras clínicas ou ambientais, onde o objetivo é avaliar a diversidade de microrganismos e identificação de disbioses presente na amostra. Com o Varsmetagen, é possível comparar os resultados com o banco de referência do Human Microbiome Project (HMP), além de inspecionar métricas de qualidade, observar disbioses e obter representações gráficas de abundância e diversidade do microbioma (bactérias e fungos).
Metagenômica
O pipeline de bioinformática para metagenomas possibilita a detecção de diversos patógenos (bactérias, vírus, fungos e parasitas eucarióticos) e também de microrganismos simbióticos, por meio de análises taxonômicas de reads e contigs, recuperação de genomas de metagenomas (MAGs) e mapeamento direcionado em genoma de referência. Com o Varsmetagen é possível realizar o diagnóstico de doenças infecciosas, caracterizar o microbioma e micobioma, e emitir laudos e relatórios completos sobre os resultados.
Como o Varsmetagen atua?
Nossa solução baseada em nuvem permite que pesquisadores e profissionais da saúde investiguem dados genéticos de microrganismos de forma ágil, eficiente e escalável.
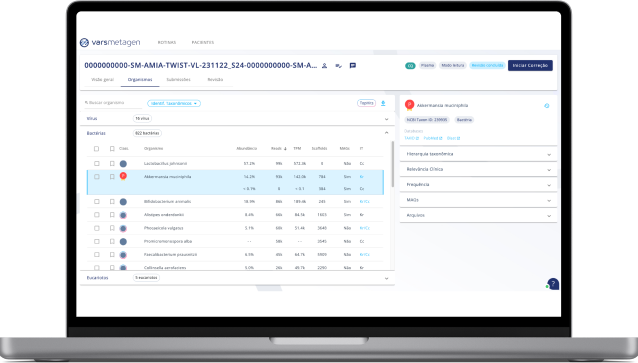
1. Ferramentas gráficas
Nossa solução baseada em nuvem permite que pesquisadores e profissionais da saúde investiguem dados genéticos de microrganismos de forma ágil, eficiente e escalável.

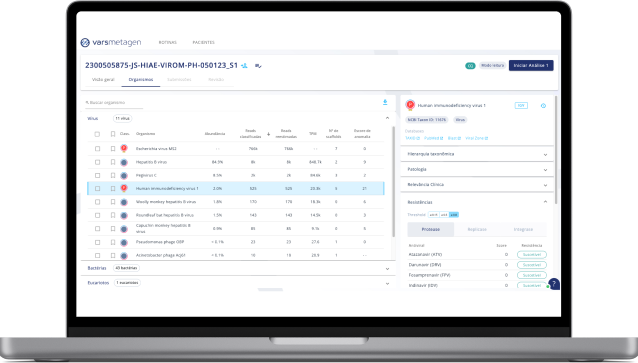
2. Anomaly meter
Algoritmos de Inteligência Artificial desenvolvidos de maneira exclusiva para fornecer escores de anomalia aos usuários, permitindo distinguir microrganismos da microbiota normal e agentes etiológicos de forma rápida e eficaz.

3. Interface intuitiva
Nossas ferramentas em estado da arte foram desenvolvidas com diferentes tipos de usuários em mente. Nossos usuários não precisam de conhecimentos avançados em programação para realizar análises.

4. Segurança
O Varsmetagen utiliza protocolos de segurança de primeira linha, em acordo com a Lei Geral de Proteção de Dados (LGPD) e com as mais modernas práticas de segurança de dados adotadas pelo Hospital Israelita Albert Einstein. Dados submetidos à plataforma são protegidos pela nossa política de privacidade e são de propriedade da instituição que fez a submissão.

Cases de Sucesso
Projeto Sars-Omics
O Projeto SarsOmics, financiado pelo CNPq, surgiu no início da pandemia e realiza o sequenciamento de vírus da COVID-19, o SARS-CoV-2, além de estudar coinfecções com outros vírus, bactérias e fungos no trato respiratório de pacientes com COVID-19. Uma quantidade gigantesca de dados NGS vem sendo gerada pelo projeto, e a praticidade e escalabilidade do Varsmetagen tem ajudado no processo de análise e interpretação destes resultados.

Cursos e treinamentos em medicina de precisão de doenças infecciosas
Apresentação Deyvid Amgarten
Bioinformata Varsomics
Apresentação Ana Carolina Soares
Bioinformata Varsomics
Apresentação Raquel Riyuzo
Bioinformata Varsomics
Programa de Early Users




IA integrado a um datalake metagenômico
Varsmetagen é uma plataforma baseada nas mais avançadas tecnologias de computação na nuvem, o que permite a criação de um data-lake metagenômico para análise integrada de dados ômicos das instituições que desejarem usufruir desta tecnologia. Através do data-lake é possível estruturar dados de diferentes tipos e origens, produzindo tabelas e repositórios com o dado centralizado para análises estatísticas, treinamento de modelos de inteligência artificial e visualizações de dashboards. Muitas das análises possibilitadas pelo data-lake se tornaram features disponíveis a todas as organizações que utilizam o Varsmetagen, como o Anomaly meter™ e o Varsdata™.
Conheça a nossa equipe de bioinformatas
Deyvid Amgarten, PhD
Metagenômica
e Genômica viral
Ana Carolina Soares, PhD
Metagenômica
e Microbiologia
Raquel
Castillo, PhD
Metagenômica e genômica bacteriana
Raquel
Riyuzo, PhD
Metagenômica
e Genômica viral
Erick
Dorlass, MSc
Genômica
Viral
Tania Girão Mangolini
Sistemas em Bioinformática
Pedro
Sebe
Ciência de Dados
e Estatística

Quem somos nós:
A Varsomics pertence ao laboratório clínico do Einstein. Somos especialistas em bioinformática com serviços aplicados ao diagnóstico clínico e pesquisas acadêmicas. Nossas principais soluções são o Varstation, plataforma para processamento de variantes genéticas humanas, e o Varsmetagen, plataforma para identificação de microrganismos a partir de dados de sequenciamento.Nossa Missão:
Viabilizar e promover o acesso e uso de novas tecnologias genéticas para aprimorar tratamentos médicos e a saúde da população.Publicações científicas
João N. de Almeida Jr, André Mario Doi, Maria Julia L. Watanabe, Maira Maraghello Maluf, Cecília Leon Calderon, Moacyr Silva Jr, Jacyr Pasternak, Paula Célia M. Koga, Kelly Aline S. Santiago, Luis Fernando C. Aranha, Gilberto Szarf, Gustavo B. da Silva Teles, Renée Zon Filippi, Vitor Ribeiro Paes, Marina Baeta, Nelson Hamerschlak, Cristovão Luis P. Mangueira, Marines Dalla Valle Martino
DOI
10.1111/
myc.13433
DOI
10.1136/
pn-2023-003795
DOI
10.1016/
j.spinee.2021.09.005
Fernanda de Mello Malta, Deyvid Amgarten, Ana Catharina de Seixas Santos Nastri, Yeh-Li Ho et al.
DOI
10.3201/
eid2606.200099
Roberta Cardoso Petroni, Anelisie da Silva Santos, Marcio Anunciação Menezes, Ana Paula Moreira Salles et al.
DOI
10.1016/
j.bjid.2021.102199
Marina Barrionuevo Mathias, Fernando Gatti Menezes, Gustavo Bruniera Peres Fernandes, Vitor Ribeiro Paes et al.
DOI
10.1212/
CPJ.0000000000200167
Lia Cunha, Adriana Luchs, Lais S. Azevedo, Vanessa C. M. Silva et al.
DOI
10.3390/
v15020335


